趣味のゲノム解析に成功!【追記あり】 [ゲノム解析で古代史]
前回の続きです。
試行錯誤の末、意外にあっさりと成功しました!
大変お世話になったのは↓のサイトです。
バイオインフォマティクスでゲノムワイド関連解析(GWAS)

ありがとうございま~す。
本当に素晴らしいです![[exclamation]](https://blog.ss-blog.jp/_images_e/158.gif)
![[1]](https://blog.ss-blog.jp/_images_e/125.gif) plinkを使ってゲノムの集団構造を体験する-パート1
plinkを使ってゲノムの集団構造を体験する-パート1
![[2]](https://blog.ss-blog.jp/_images_e/126.gif) plinkを使ってゲノムの集団構造を体験する-パート2
plinkを使ってゲノムの集団構造を体験する-パート2
試しに14番染色体をプロットしてみましたが、結果はこのサイトの1番染色体とほとんど同じでした。
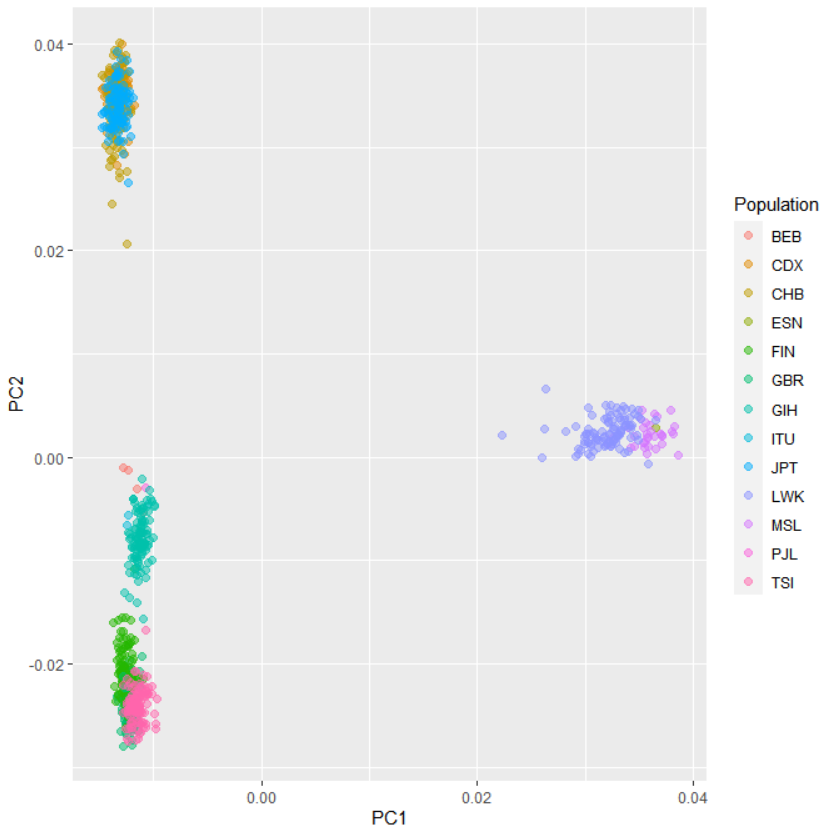
なお、私は慣れたところでRを遣い、これに関連して3箇所ほど修正しました。
①4を、data <- read.delim('integrated_call_samples_v3.20200731.ALL.ped', header=TRUE) に変更
②パッケージ ggplot2 をインストール
③5の1行目を、columnList <- c("Individual.ID","Population") に変更 【追記1 Population表示が正しくなるよう修正】
どうやら、パソコンのメモリは8GBだと足りないようです。
このサイトのように、主成分を10個指定すると、私のパソコンではメモリ不足になってしまいました…![[たらーっ(汗)]](https://blog.ss-blog.jp/_images_e/163.gif)
CPUは、それほどは高性能でなくともいいらしく、私はi5-7500なのですが…計算自体は数分で終わりました。![[手(チョキ)]](https://blog.ss-blog.jp/_images_e/87.gif)
ただ、ゲノムデータは1染色体ほぼ1GBあるので、高速回線が必須となります。
それにしても、個人レベルで気軽にゲノム解析ができるようになったのだから、世の中も随分と変わったものです。
参考までに、ソフトとデータの入手はすべて無料です。
ただし、プライバシーの関係で、センシティブなデータには厳重な手続きが必要となります。
【追記2】
これで、血液型別にゲノム解析をして、もしも差が出たら万々歳です![[るんるん]](https://blog.ss-blog.jp/_images_e/146.gif)
試行錯誤の末、意外にあっさりと成功しました!
大変お世話になったのは↓のサイトです。
バイオインフォマティクスでゲノムワイド関連解析(GWAS)
ありがとうございま~す。
本当に素晴らしいです
試しに14番染色体をプロットしてみましたが、結果はこのサイトの1番染色体とほとんど同じでした。
なお、私は慣れたところでRを遣い、これに関連して3箇所ほど修正しました。
①4を、data <- read.delim('integrated_call_samples_v3.20200731.ALL.ped', header=TRUE) に変更
②パッケージ ggplot2 をインストール
③5の1行目を、columnList <- c("Individual.ID","Population") に変更 【追記1 Population表示が正しくなるよう修正】
どうやら、パソコンのメモリは8GBだと足りないようです。
このサイトのように、主成分を10個指定すると、私のパソコンではメモリ不足になってしまいました…
CPUは、それほどは高性能でなくともいいらしく、私はi5-7500なのですが…計算自体は数分で終わりました。
ただ、ゲノムデータは1染色体ほぼ1GBあるので、高速回線が必須となります。
それにしても、個人レベルで気軽にゲノム解析ができるようになったのだから、世の中も随分と変わったものです。
参考までに、ソフトとデータの入手はすべて無料です。
ただし、プライバシーの関係で、センシティブなデータには厳重な手続きが必要となります。
【追記2】
これで、血液型別にゲノム解析をして、もしも差が出たら万々歳です
2022-02-27 18:33
コメント(0)




コメント 0