趣味でゲノム解析をする方法 [ゲノム解析で古代史]
自分でゲノム解析をする場合の備忘録です。
下は、国立科学博物館の例ですが、ソフトはPLINKを使っているようです。
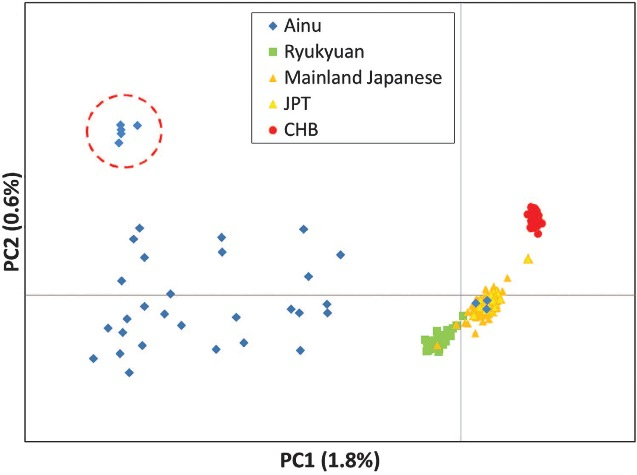
なにはともあれ、相当高性能のパソコンが必要で、メモリは最低でも16GBが必須となります。
HDDも500GBでは足りないかもしれません。
非力なパソコンだと、処理に時間がかかったり、メモリが足りなくてエラーになりやすく、相当イライラします。
さて、ゲノムのデータについては、無料で1000人ゲノムプロジェクトから入手可能です。
入手先と入手方法はこちら
↓
https://www.internationalgenome.org/data-portal/sample
https://biotech-lab.org/articles/6385#i-2
Data types は Variatnts(VCFファイル)を選択します。
注意点ですが、Filter by populationで選択しても、SGDP以外は調査対象者全員分が付いてくるので、誰を選択しても同じ内容です。
また、基本的にファイルは染色体別なので、ALL.chr○…は全部ダウンロードする必要があります。
容量は、1つのファイルは圧縮済み(gzフォーマット)でも数100GB以上なので、トータルでは結構なサイズとなります。
基本的にFTPでダウンロードするので、Windowsならコマンドラインか専用クライアントソフトが必要です。
なお、最近のソフトは圧縮済みでもそのまま取り扱えるようになったので、ちょっと便利になりました。
日本人の1094人のデータ
↓
https://seichannel.net/2020/04/13/snp_analysis_for_japanese_data_for_begginers/
次に分析方法ですが、Rを使う方法とPLINKを使う方があります。
Rを使う方法
↓
① https://www.iu.a.u-tokyo.ac.jp/lectures/AG16/180509/20180509.pdf (PDF)
② https://nemunemu-nyanko.hatenablog.com/entry/2020/12/19/191440#google_vignette
pcaMethodsのインストール方法
↓
https://www.kkaneko.jp/data/rna/velocyto.html (ページの一番下)
PLINKを使う方法
↓
① http://www.sg.med.osaka-u.ac.jp/files/StatGenSummerSchool2021_GenomeDataAnalysis1.pdf (PDF)
② https://qiita.com/insilicomab/items/790553a5a92d759c6ab3
まだ、主成分分析まで完了していないのですが、頑張りたいと思いますv(^^)。
下は、国立科学博物館の例ですが、ソフトはPLINKを使っているようです。

なにはともあれ、相当高性能のパソコンが必要で、メモリは最低でも16GBが必須となります。
HDDも500GBでは足りないかもしれません。
非力なパソコンだと、処理に時間がかかったり、メモリが足りなくてエラーになりやすく、相当イライラします。
さて、ゲノムのデータについては、無料で1000人ゲノムプロジェクトから入手可能です。
入手先と入手方法はこちら
↓
https://www.internationalgenome.org/data-portal/sample
https://biotech-lab.org/articles/6385#i-2
Data types は Variatnts(VCFファイル)を選択します。
注意点ですが、Filter by populationで選択しても、SGDP以外は調査対象者全員分が付いてくるので、誰を選択しても同じ内容です。
また、基本的にファイルは染色体別なので、ALL.chr○…は全部ダウンロードする必要があります。
容量は、1つのファイルは圧縮済み(gzフォーマット)でも数100GB以上なので、トータルでは結構なサイズとなります。
基本的にFTPでダウンロードするので、Windowsならコマンドラインか専用クライアントソフトが必要です。
なお、最近のソフトは圧縮済みでもそのまま取り扱えるようになったので、ちょっと便利になりました。
日本人の1094人のデータ
↓
https://seichannel.net/2020/04/13/snp_analysis_for_japanese_data_for_begginers/
次に分析方法ですが、Rを使う方法とPLINKを使う方があります。
Rを使う方法
↓
① https://www.iu.a.u-tokyo.ac.jp/lectures/AG16/180509/20180509.pdf (PDF)
② https://nemunemu-nyanko.hatenablog.com/entry/2020/12/19/191440#google_vignette
pcaMethodsのインストール方法
↓
https://www.kkaneko.jp/data/rna/velocyto.html (ページの一番下)
PLINKを使う方法
↓
① http://www.sg.med.osaka-u.ac.jp/files/StatGenSummerSchool2021_GenomeDataAnalysis1.pdf (PDF)
② https://qiita.com/insilicomab/items/790553a5a92d759c6ab3
まだ、主成分分析まで完了していないのですが、頑張りたいと思いますv(^^)。
2022-02-26 22:13
コメント(0)




コメント 0